La vida, en su asombrosa complejidad, se despliega a través de la acción de innumerables «máquinas moleculares»: las proteínas. Estos elementos son las verdaderas obreras de nuestras células, esenciales para casi todas sus funciones. Construyen nuestras estructuras, transportan oxígeno, defienden nuestro organismo, catalizan las reacciones que nos mantienen vivos y transmiten las señales que nos coordinan. Cada proteína tiene una tarea específica, y esa tarea viene determinada por su forma tridimensional única, como una llave precisa para una cerradura concreta.
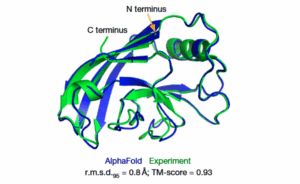
Comparación entre la estructura proteica predicha por AlphaFold (azul) y la determinada experimentalmente (verde), mostrando una alta concordancia. Fuente: Highly accurate protein structure prediction with AlphaFold John Jumper, Richard Evans , Alexander Pritzel y otros.
Pero aquí radicaba uno de los mayores enigmas de la biología durante más de medio siglo: si conocemos la «receta» de una proteína (su secuencia de aminoácidos, dictada por nuestro ADN), ¿cómo podemos saber qué forma adoptará? Averiguar esta estructura 3D era un proceso arduo. Los métodos experimentales, aunque potentes, son lentos, caros y no funcionan para todas las proteínas. Imaginen intentar entender cómo funciona una máquina complejísima sin poder ver claramente sus piezas. Este «problema del plegamiento de proteínas» representaba una barrera fundamental para comprender a fondo la vida y las enfermedades.
En este escenario, la inteligencia artificial (IA) ha irrumpido como una fuerza transformadora. En particular, AlphaFold, un sistema desarrollado por DeepMind (empresa de Google), ha marcado un antes y un después. Utilizando técnicas avanzadas de aprendizaje profundo, AlphaFold ha demostrado una capacidad asombrosa para predecir la forma 3D de las proteínas con una precisión que, en muchos casos, rivaliza con los resultados de laboratorio. ¿Cómo lo logra y qué puertas está abriendo este avance para la ciencia y la medicina?
El laberinto del plegamiento: comprendiendo el reto
Una proteína nace como una larga cadena de aminoácidos. Para ser funcional, esta cadena debe plegarse sobre sí misma de una manera muy específica y compleja, formando hélices, láminas y recovecos tridimensionales. Primero, la cadena se pliega en tramos cortos creando patrones regulares, como espirales (llamadas hélices alfa) o superficies planas plegadas (láminas beta); esto se llama estructura secundaria. Luego, estas hélices y láminas, junto con el resto de la cadena, se organizan en el espacio para dar a la proteína su forma tridimensional global única, conocida como estructura terciaria. Finalmente, algunas proteínas consisten en varias cadenas ya plegadas que se ensamblan, formando una estructura cuaternari. Este plegamiento no es aleatorio; está guiado por las interacciones químicas entre los aminoácidos. Sin embargo, el número de formas posibles que podría adoptar una cadena es astronómico, tanto que, si una proteína probara todas las opciones, tardaría más que la edad del universo en encontrar la correcta (la Paradoja de Levinthal). Y, sin embargo, en nuestras células, lo hacen en segundos.
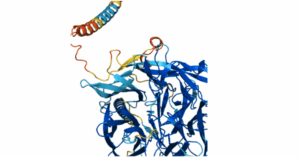
Visualización de la estructura tridimensional de una proteína, predicha por la inteligencia artificial AlphaFold. Programas como este permiten a los científicos ‘ver’ la forma compleja que adoptan las proteínas (mostrando aquí hélices en espiral y láminas planas), lo cual es fundamental para entender cómo realizan sus funciones vitales en nuestro organismo. Fuente: Base de datos de estructura de proteínas AlphaFold Desarrollado por Google DeepMind y EMBL-EBI
Durante décadas, los científicos usaron técnicas como la cristalografía de rayos X o la crio-microscopía electrónica para «fotografiar» estas estructuras. Son técnicas poderosas pero con limitaciones: requieren mucha proteína purificada, a veces necesitan cristalizarla (un proceso difícil y no siempre posible), y son costosas y lentas. Como resultado, conocíamos la secuencia de millones de proteínas, pero solo teníamos la estructura detallada de una pequeña fracción. Métodos computacionales anteriores intentaron predecir la estructura, pero su precisión era limitada, especialmente para proteínas sin «parientes» conocidos.
AlphaFold: la IA que aprendió a «ver» la estructura
AlphaFold representa un cambio de enfoque radical. Utiliza redes neuronales profundas, inspiradas en el funcionamiento del cerebro, que aprenden patrones complejos a partir de enormes cantidades de datos. ¿De dónde vienen esos datos? Principalmente de dos fuentes públicas:
- La Protein Data Bank (PDB): el archivo mundial que contiene cientos de miles de estructuras de proteínas determinadas experimentalmente por científicos durante décadas.
- Bases de datos de secuencias: gigantescos repositorios con millones de secuencias de proteínas de todos los seres vivos conocidos.
Entrenándose con estos datos, AlphaFold aprendió las intrincadas reglas que conectan la secuencia de una proteína con su forma 3D final. Utiliza arquitecturas sofisticadas (como mecanismos de atención, similares a los usados en IA de lenguaje) para considerar cómo interactúan entre sí aminoácidos que pueden estar muy separados en la cadena lineal. El sistema no solo aprende de los datos, sino que también incorpora principios biofísicos sobre cómo se pliegan las proteínas.
El momento decisivo llegó en la competición internacional CASP14 (2020), una evaluación «a ciegas» donde los sistemas computacionales predicen estructuras aún no publicadas. AlphaFold 2 demostró una precisión asombrosa, superando ampliamente a todos los demás métodos y alcanzando niveles comparables a los experimentales para muchas proteínas. Fue aclamado como un hito que abría una nueva era.
Un atlas molecular para todos: democratizando el acceso
Tan importante como el avance técnico fue la decisión de DeepMind, en colaboración con el Instituto Europeo de Bioinformática del EMBL (EMBL-EBI), de hacer públicas las predicciones. Crearon la AlphaFold Protein Structure Database (AlphaFold DB), una base de datos online y gratuita.
Facilitando el acceso. Cuatro maneras de acceder a las estructuras de proteínas predichas en la AFDB. En sentido horario desde arriba a la izquierda: sitio web, API, BigQuery y descarga FTP. Fuente: AlphaFold, Protein Structure Database, Developed by Google DeepMind and EMBL-EBI
Inicialmente lanzada con estructuras del proteoma humano y otros organismos clave [100], se expandió masivamente en 2022 para incluir más de 200 millones de predicciones estructurales, cubriendo casi todas las proteínas conocidas por la ciencia, desde bacterias hasta plantas y animales. Cada entrada incluye no solo la estructura 3D predicha, sino también indicadores de confianza (llamados pLDDT y PAE) que permiten a los científicos saber qué partes del modelo son más fiables.
Este gesto de ciencia abierta ha tenido un impacto inmenso. Investigadores de todo el mundo, sin necesidad de costosos equipos de laboratorio, pueden ahora obtener un modelo estructural de alta calidad para su proteína de interés en cuestión de minutos. AlphaFold DB se ha convertido en una herramienta fundamental utilizada por millones de científicos, acelerando la investigación en innumerables campos.
Acelerando descubrimientos: nuevas vías para la investigación
La disponibilidad de este «atlas» estructural está catalizando la ciencia de formas muy diversas, aunque es crucial recordar que son predicciones que a menudo requieren validación experimental:
- Hacia nuevos fármacos: conocer la forma 3D de una proteína clave en una enfermedad (una bacteria resistente, un virus, una célula cancerosa) es esencial para diseñar fármacos que la bloqueen. AlphaFold acelera enormemente este proceso: ayuda a identificar posibles «dianas» terapéuticas, y permite usar ordenadores para diseñar y probar virtualmente moléculas que encajen en esas dianas. Se está explorando activamente su uso contra la resistencia a antibióticos, la malaria, enfermedades virales o el cáncer. La versión más reciente, AlphaFold 3, incluso intenta predecir directamente cómo interactúan las proteínas con posibles fármacos.
- Comprendiendo enfermedades: muchas enfermedades genéticas se deben a mutaciones que alteran la estructura de una proteína. AlphaFold permite visualizar cómo una mutación podría afectar al plegamiento o función de la proteína, ayudando a entender por qué causa la enfermedad (desde la fibrosis quística a enfermedades neurodegenerativas). Esto puede mejorar el diagnóstico y guiar terapias personalizadas.
- Impulsando la biotecnología: las enzimas (proteínas que aceleran reacciones) son herramientas clave en la industria. La ingeniería de proteínas busca crear enzimas a medida para producir fármacos de forma más limpia, desarrollar biocombustibles, o incluso degradar plásticos. AlphaFold facilita el diseño de estas nuevas enzimas al proporcionar su estructura como punto de partida.
- Avances en biología fundamental: para miles de proteínas cuya función era un misterio, su estructura predicha por AlphaFold ofrece pistas cruciales. Comparando su forma con la de proteínas conocidas, los científicos pueden inferir qué podrían estar haciendo en la célula. También permite estudiar la evolución de las proteínas a través de las distintas especies como nunca antes.
El horizonte científico: la IA como compañera indispensable
AlphaFold es un ejemplo importante, pero la IA está transformando muchas otras áreas científicas: desde analizar genomas, descubrir nuevos materiales, modelar el clima o analizar datos del universo. Todo apunta a que se está consolidando como una nueva forma de hacer ciencia, complementando la teoría y la experimentación.
El futuro de la biología estructural probablemente implique una integración aún mayor entre la IA y los experimentos. Las predicciones guiarán los experimentos, y los datos experimentales ayudarán a refinar y mejorar los modelos de IA. Todavía hay retos: predecir cómo se mueven las proteínas (dinámica), cómo interactúan en grandes complejos, o el efecto exacto de las mutaciones. Pero el camino está trazado.
Conclusión: una nueva era de conocimiento molecular
AlphaFold está demostrando de forma relevante el poder de la inteligencia artificial para investigar desafíos científicos fundamentales y acelerar el descubrimiento. No se trata de reemplazar a los científicos, sino de potenciar su ingenio, proporcionándoles herramientas para explorar la complejidad de la vida a un nivel de detalle y a una velocidad inimaginables hace solo unos años.
Al descifrar la estructura de las proteínas, la IA nos está ayudando a entender el «código de la vida» de una forma más profunda. Esta nueva era de conocimiento molecular, impulsada por la sinergia entre la inteligencia humana y la artificial, no solo amplía las fronteras de la ciencia, sino que también nos ofrece la esperanza de encontrar soluciones innovadoras para grandes retos en salud y sostenibilidad global.